Hensikt
Sikrer at identifisering av mikroorganismer ved hjelp av MALD I-TOF (Matrix Assisted Laser Desorption/Ionization Time-Of-Flight) blir utført på en korrekt måte.
Omfang
Alle bioingeniører som er opplært til å utføre denne type analysearbeid ved Lab. for med.mikrobiologi.
Grunnlagsinformasjon
ANALYSEPRINSIPP OG TERMINOLOGI
MALDI -TOF (Matrix Assisted Laser Desorption / Ionization Time-Of-Flight) massespektrometri kan benyttes til identifikasjon av kulturer av bakterier og sopp, både i renkultur og enkeltkolonier i en blandingskultur. Metoden kan erstatte eksisterende identifikasjonssystemer.
MALDI -TOF massespektrometri koblet sammen med MALDI Biotyper programvare er et identifikasjonssystem som er automatisk og gir hurtig identifikasjon av mikroorganismer uten å være avhengig av andre fenotypiske parametre. Systemet baserer seg på undersøkelse av proteiner, hovedsakelig ribosomale proteiner, ved hjelp av massespektrometri.
Prinsipp
Et lite volum av ren bakterie- eller soppkultur avsettes på en metallplate og dekkes med en matriks. Deretter bestråles matriksen med laserenergi slik at mikrobielle proteiner ioniseres. Disse ionene blir så aksellerert i et magnetisk felt, og ved å måle tiden partiklene benytter på flukten (Time of Flight) gjennom det magnetiske feltet kan størrelsen på partiklene beregnes. Ribosomale proteiner vil danne massespekter karakteristisk for ulike bakterie- eller sopp-arter, og slike spekter kan sammenlignes med spektra i databaser med kjente bakterie- og sopparter for identifikasjon
REFERANSER
Hovedreferanse:
(1) MBT Compass 2023 v.13
(2 Microflex User Manual rev. E april 2014
(3) MALDI Mass Spectrometry, july 04
(4) Bruker Daltonics
(5) Improved Performance of Bacterium and Yeast Identification by a Commercial Matrix-Assisted Laser Desorption Ionization–Time of Flight Mass Spectrometry System in the Clinical Microbiology Laboratory, John Haigh, Amarjeet Degun, Melvyn Eydmann, Michael Millar, Mark Wilks, Journal of Clinical Microbiology
(6) Pakningsvedlegg ligger på teams: Pakningsvedlegg og mottakskontroll
Offisielle godkjenninger: IVD, CE
Eksterne valideringer: St. Olavs Hospital med 200 bakteriestammer, 2012.
INTERN KVALITETSKONTROLL
Kvalitetskontroll
- 1,0 μl av Bruker BTS (BTS; E.coli DH5α) settes opp som intern kontroll en gang pr.dag. Oppbevares i fryseskap på pusslaben.
- Resultatene føres på vedlikeholdsskjemaet for MaldiTof. Godkjent positiv kontroll ved score ≥ 2.0, negativ ved no peaks found.
Kontrolløsninger/referansematerialer
MALDI -TOF kontroll (BTS- Bacterial Test Standard, pos.kontroll)
Tillaging av MALDI-TOF kontroll
- Ta opp et BTS-rør fra fryseren (gul topp) Nok til ca. 40 tester
- Bruk ny OS (OS=Standard Solvent). Pipetter 50 µL OS
- Bland med pipetten opp/ned minst 20 ganger. NB! Unngå å skumme
- Inkuber i 5 min ved romtemperatur
- Repeter punkt 3
- Sentrifuger 2.min. ved 13000 rpm
- Fordel 3-4 µL i eppendorfrør.
- Frys ned ved minst 18 ºC, fryseskap på pusslaben.
- Før opp lot og dato for oppløsning på fryseboks.
- Kjør en kontroll med den nye BTS’en
- Signer på vedlikeholdsskjema.
EKSTERN KVALITETSKONTROLL
Med. mikrobiologi er med i ringtestprogrammet til Folkehelseinstituttet og Neqas.
Arbeidsbeskrivelse
ANSVAR
Fagbioingeniør som er tillagt denne analysegruppen, har ansvar for vedlikehold av denne prosedyren. Superbrukere har ansvar for feilsøking og kontakt med servicetekniker. Opplærte bioingeniører har ansvar for kalibrering, restart av pc, source clean og detector check se ALM-MM-Bakt; MaldiTof Microflex vedlikehold.
HMS/sikkerhetsinformasjon
Bruk spesialhansker ved tillaging av løsninger. Bruk spesialhansker og avtrekk ved arbeid med maursyre.
Stammer som ikke kan identifiseres på MaldiTof
Inaktiveringsrutiner for MALDI-TOF inaktiverer ikke sporer. En må derfor ikke utføre denne metoden ved mistanke om B. anthracis. Det er heller ikke mulig å skille B. anthracis fra andre andre agens i B. cereus gruppen ved massesepektrometri. Motsatt vei, så kan B. cereus, eller andre medlemmer av gruppen, også gi resultatet «påvist B. anthracis» ved analyse. Det er altså essensielt å være sikker på at det ikke er miltbrann som har gjort pasienten syk før man kjører denne metodikken. Videre skal bare hemolytiske kolonier av Bacillus spp. testes på MaldiTof.
ARBEIDETS GANG
Reagenser og klargjøring av disse
OS Organic solution (Standard Solvent)
475 µl destillert/sterilt vann i et rør
500µl Actetonitrile (ACN)
25µl 100% Trifluoracetic Acid (TFA)
Vortex
Holdbarheten til OS er 7-10 dager. Skriv på røret tillagingsdato.
Matrix
- Ta fram 1 rør med ”HCCA matrix portioned” (Oppbevares på kjølerom RTB 024)
- Tilsett 250 μl ”OS” til et rør ”HCCA matrix portioned” og vortex til all matrixkrystaller er oppløst
- Merk røret med dato og signatur, og signer på skjema
- Ferdig laget matrix kan oppbevares i romtemp.i opptil 2 uker (”best før”) .Oppbevares mørkt
MALDI-target (Metallplate med 96 posisjoner)
Vi har to typer MALDI target plate, en gjenbrukbar target plate (polert stål) og en engangstarget plate med 48 posisjoner. Hver target plate har en egen ID, denne brukes som gjenkjenning av type target plate som er i bruk for MALDI Biotyper. Gjenbruks target plate har 101 XXXXXXX. Engangstarget plate har et unikt nummer for registrering i MBT compass. Disse ligger i skapet til høyre under benken som Maldien står på. Holdere for engangstarget er også der.
Rene target ligger i grå boks merket ”RENE”
Target til vasking legges i grå boks merket ”URENE”
Prøvepreparering (DT=Direkte transfer)
- 1 enkelt koloni (helst ikke mer enn 24-48 t gammel) gnies som en tynn film direkte på MALDI target. Bruk en tannpirker
- Lufttørk
- Det er bedre å bruke lite materiale enn for mye
- 1,0 μl matrix løsning legges over straks det er tørt (innen 1 time)
- Lufttørk
- Target er klar til å kjøres på MALDI-TOF
Analyseutstyr og klargjøring av disse
Kalibrering gjøres en gang pr.uke med BTS
- Ansvar: Alle som utfører vedlikeholdsoppgaver
- MALDI-target med dagens positive kontroll settes i maskina
- Åpne Flex-kontroll
- Finn posisjon for kontrollen
Trykk ”Calibrate”Etter noen sekunder kommer resultatet om kalibreringen er godkjent
Kvitér på vedlikeholdsskjema
Vasking av MALDI – target
Arbeid foregår i avtrekksskap med hansker
- Tom petriskål dekkes med linsepapir
- MALDI-target (platen) legges oppi
- Dekk over med 75 % sprit/etanol. La stå i 5 min.
- Tørk av med linsepapir
- Skyll platen under varmt springvann
Bruk tommel til å stryke over targets mens vannet renner - Tørk av med linsepapir
- Dekk over med 75 % sprit. La stå i 3 min.
- Tørk av med linsepapir
- Skyll platen under varmt springvann
- Bruk tommel til å stryke over alle targets mens vannet renner
- Tørk av med linsepapir
- Deponer 150µl 80 % Trifluoreddik syre (TFA) på hver plate. Bruk bomullspinne til å gni godt over alle targets
- Tørk av med linsepapir
NB! Ikke få TFA på hanskene - Skyll godt med destillert vann og tørk av med linsepapir. Tørk godt av på undersiden av platen med linsepapir. Viktig å skylle godt med destillert vann da mineraler og ioner i springvann kan forstyrre under avlesning.
- Legg platen oppi lagringsboksen
La boksen stå åpen 5-7 min før lokket lukkes
Platen er klar til bruk neste dag
Usikkerhetsbidrag, feilkilder og interferens
- For tynn/tykk spot
- For gammel matrix
- Ikke godt nok oppløst matrix
- Targetplate uren
Fremgangsmåte/Utførelse av analysen
Oppstart MaldiTof pc
- Dobbelklikk på Flex Controll
- Dobbelklikk på MBT Compass. MBT Compass vil åpnes
|
Gå til Epicenter Trykk på knapp nr. 3 fra høyre i øverste fanerekka |
|
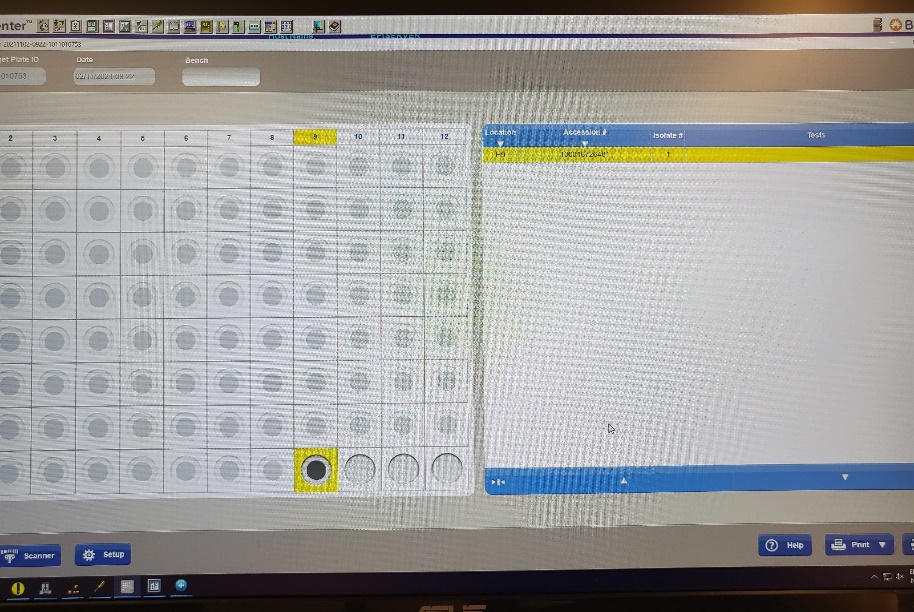
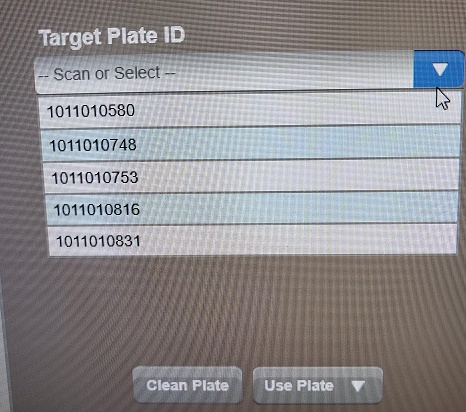
For plate i bruk:
- Scan barkoden på skåla med mikroben du skal identifisere. Det vil være neste ledige posisjon og er merket gul.
- Gni mikroben på plata og tilsett matrix. Når matrix er tørr, gå til MaldiTof
- Trykk på Grønn knapp ( IN / OUT )
- Sett inn target plate
- Tørk O-ringen (svart gummi) med en finger
- Lukk forsiktig igjen lokket
- Trykk på Grønn knapp ( IN / OUT )
På MaldiTof pc
· Trykke Start acquisition i MBT Compass
MaldiTof starter når tilstrekkelig vakuum er oppnådd.
- For utskrift av analyserapporten trykk på Report når analysen er ferdig
- Trykk på skriver ikonet i PDF filen som åpnes. I feltet pages → skriv antall sider som skal skrives ut
- Trykk print. Lukk siden etterpå.
På MaldiTof
Trykk på Grønn knapp (IN / OUT) og ta ut target plate
- Trykk på Grønn knapp på nytt (lokket blir stengt )
- Avslutt MBT Compass ved å trykke på det røde krysset
- Avslutt Flex Control ved å trykke på det røde krysset
- Bekreft avslutning ved å svare Yes
Ved oppstart av ny plate
På Epicenter
|
Da er platen klar til bruk
Ved oppsett av pos/neg kontroll velges det fra bildet som kommer opp når en trykker use plate. |
|
Vurdering
Tolking av resultater
|
Score |
Kategori |
Vurdering |
Fargekode |
|
2,300-3,000 |
+++ |
Identifikasjon på sikkert species nivå |
Grønn |
|
2,000-2,299 |
++ |
Identifikasjon på species nivå |
Grønn |
|
1,700-1,999 |
+ |
Identifikasjon på genus nivå |
Gul |
|
0,000-1,699 |
0 |
Ingen sikker identifikasjon |
Rød |
· Alle identifikasjoner med minst én ID score > 2,0 er godkjent ID
NB! Ved Neisseria meningitidis må MALDI-TOF resultat bekreftes med sukkerforgjæring og/eller N. meningitidis PCR. Vi må sende stammen til SOH. Blodkultur og spinalvæske gis ut med sannsynlig Neisseria meningitidis, men med kommentar om at stammen er sendt SOH. For annet prøvemateriale gis ut Neisseria spp. og at stammen er sendt SOH.
• ID score > 1,7: For alle identifikasjoner med ID score >1,7:
1. Bakterier: Alle identifikasjoner med ID score > 1,7 (men < 2,0) er godkjent ID dersom man har annen informasjon om isolatet som f.eks. cellemorfologi, cellevegg, enkle fenotypiske reaksjoner, agglutinasjon, osv. Vurder besvaring av identifikasjon på kun genusnivå i hvert tilfelle (f.eks. Corynebacterium sp.). Isolat fra blodkultur og spinalvæske skal aldri besvares på kun genusnivå. For andre sterile materialer bør lege konfereres. Dersom minst fire av de først foreslåtte identifikasjoner på en spot viser samme species, forsterkes sannsynligheten for korrekt artsidentifikasjon.
2. Sopp: Alle identifikasjoner med ID score > 1,7 (men < 2,0) er godkjent ID dersom begge spotene viser samme identifikasjon.
• ID score < 1,7: Alle identifikasjoner med ID score < 1,7 er ikke godkjent ID. Dersom resultatet er < 1,7 (kommer i kategori 0), ekstraheres prøven og analyseres på nytt. Dersom forenklet ekstraksjon gir heller ikke noen ID, vurder å utføre etanol-maursyre ekstraksjon. I tillegg kan man utføre annet identifikasjonssystem (phoenix, etc.). Diskuter evt. videre identifisering med lege for å vurdere om prøven skal sendes til SOH til identifikasjon ved bruk av annen metodikk, f. eks. 16S rDNA sekvensering.
• Ikke skillbare mikroorganismer: Mikroorganismer som normalt sett ikke kan skilles fra hverandre ved hjelp av gensekvensering av ribosomale proteingener (16s, 18s osv.) kan generelt sett heller ikke skilles med dette identifikasjonssystemet. Dette gjelder f.eks. pneumokokker, Shigella sp. osv. Se vedlegget «Restrictions of MALDI Biotyper identification» under fanen relatert.
MaldiTof identifiserer også muggsopp. Oppsettet må gjøres med etanol/maursyreekstraksjon (ikke forenklet maursyreekstraksjon) for å hindre kontaminasjon med soppsporer til miljø og utstyr.
Omkjøring
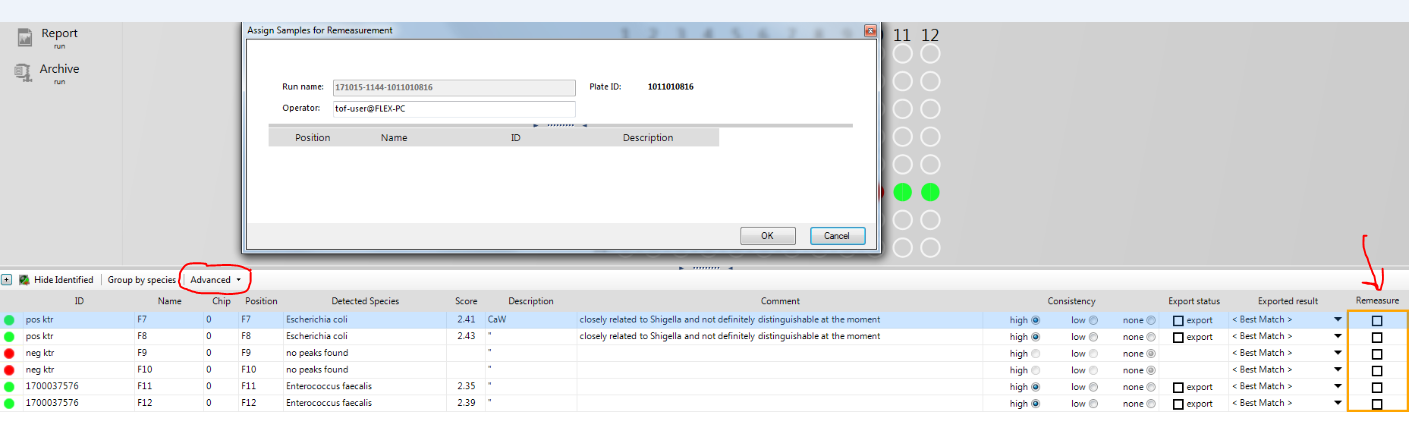
Ved behov for å kjøre på nytt en eller flere spotter, gjør som følger:
I MBT Compass, trykk på Advanced . Det vil da poppe opp bilde «Assign Samples for remeasurement». Lengst til høyre vil det komme til en kolonne «remeasure». Hak av for de nummerene du vil teste på nytt. Trykk på ok i bilde «assign samples for remeasurement».
Etanol / Maursyreekstraksjon (Formic acid)= Extended Direct Transfer (EX)
Bruk hansker
Prøvepreparering
- Pipetter 300 µl destillert H2O i eppendorf-rør
- Plukk, med øse (blank), kolonier fra stamme som skal testes og bland oppi eppendorf-røret
- Bland godt (vortex-mikser)
- Tilsett 900 µl absolutt alkohol Bland godt (vortex-mikser)
- Sentrifuger 2 min, maks hastighet, med hengslene vendt ut ( Eppendorf-sentrifuge)
- Dekanter supernatant ved bruk av steril pipette, evt. tøm forsiktig ut (Følg med på at bunnfall/pellet blir igjen)
- La topp stå åpen ei kort stund slik at rester av alkohol diffunderer ut
- Sentrifuger noen sekunder ved maks hastighet; trykk start / stopp
- Pipetter forsiktig ut alt av etanolrester (Eppendorf-pipette)
- La bunnfall tørke noen min (topp stå åpen ca.5 min / evt. i varmeskap ca.2-3 min) NB! Bunnfall må være helt tørt
- Tilsett 70 % Maursyre (Formic acid ( FA)) Vurder ut i fra hvor stor pellet er. Oppslemmet fra 1 µl øse tilsett 30 µl Evt. reduser mengde FA. Se tabell
Tabell over mengde maursyre og acetonitrile i forhold til bakteriemengde
|
|
små enkelt kolonier |
store enkelt kolonier |
1µl øse |
|
Maursyre (Formic Acid) 70% |
1-5µl |
10-20µl |
20-40µl |
|
Acetonitrile |
1-5µl |
10-20µl |
20-40µl |
- Bland godt (vortex-mikser)
- La stå i 2 min
- Tilsett lik mengde Acetonitril (AN) som FA under punkt 11
ALLTID; lik mengde FA og AN - Bland forsiktig med pipette (unngå bobler) Sentrifuger 2 min, maks hastighet
- Pipetter 1,0 µl av supernatant på target
- Lufttørk
- Tilsett 1,0 µl matrix (straks det er tørt)
- Lufttørk
- Klar til kjøring på MALDI – TOF
Forenklet maursyreekstraksjon
- 1 enkelt koloni direkte på MALDI target.
- Tilsett 1,0 μl 70% maursyre i avtrekk
- Lufttørk
- 1,0 μl matrix løsning legges over straks det er tørt (innen 1 time)
- Lufttørk
- Target er klar til å kjøres på MALDI-TOF
Ved behov for service:
Ved behov for akutt teknisk service skal henvendelsen gå til:
E mail: support.nordic@bruker.com eller på telefon: +46 8 655 25 20 (Sverige). Se også nummere oppført ved MaldiTof.
For å få tilgang til nettverksoppkobling, må Karl Andre på medisinsk teknisk kontaktes.
Vi har mulighet til å koble opp MaldiTof pc’en med Bruker via WebEx. Ikonet for dette ligger på skrivebordet øverst høyre hjørne. Når du tar dette opp, vil det stå «Enter the meeting, event or session number to join».
Du vil få et nummer fra Bruker som du legger inn her og service tekniker vil få anledning til å fjernstyre pc’en.
Back-up
Back-up av data blir lagret på Epicenter hvert døgn.